Smart Gutは、2024年7月3日より、大学や公的研究機関向けに、同社取締役CTO 服部 正平博士責任監修による「細菌叢DNA抽出サービス」を開始しました。
Smart Gut「細菌叢DNA抽出サービス」
Smart Gutは、2024年7月3日より、大学や公的研究機関向けに、同社取締役CTO 服部 正平博士責任監修による「細菌叢DNA抽出サービス」を開始!
■同社の細菌叢DNA抽出の特徴
同社が開始した細菌叢DNA抽出の受託事業は、Illumina社、PacBio社、Oxford Nanopore Technologies社の各NGSによるDNA配列シークエンシングに高い成功率と実績をもつLA法(酵素的溶菌法)をベースに独自開発した技術で運用します。
近年の研究から、LA法(酵素的溶菌法)は、N及びP法(いずれも機械的溶菌法)と高い互換性のあるヒト腸内細菌叢のDNA抽出法であることがわかりました(後述)。
注目すべきは、酵素的溶菌と機械的溶菌という溶菌メカニズムが異なる抽出法が有意に高い類似性をもつ菌種組成を互いに与えた点です。
このことはLA、N、P法から得られる菌種組成が真実の菌種組成に極めて近いことを意味します。
よって、同社が開発したDNA抽出法によって得られる細菌叢DNAからは、他方法よりも格段に正確で信頼性の高いさまざまな細菌叢解析を可能にします。
「細菌叢DNA抽出サービス」詳細:
https://www.smart-gut.com/services
■細菌叢DNA抽出法技術について
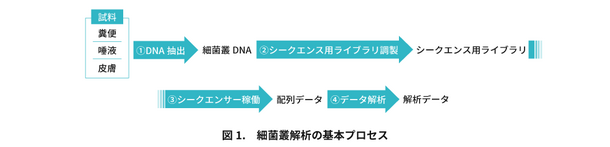
マイクロバイオームを解析する第1ステップは、試料(糞便や唾液、皮膚など)からの細菌叢DNAの抽出です。
次ぐ第2ステップは、抽出されたDNAを次世代シークエンサー(NGS)に供して、細菌叢がもつDNA配列データの取得。
そして第3ステップでは、得られたDNA配列を情報学的に解析し、菌種や菌種の遺伝子組成など一連の細菌叢データを得るに至ります(図1)。
第2ステップで得られる細菌叢のDNA配列データには2種類あり、それぞれ16SリボソームRNA遺伝子(16S)データとメタゲノムデータとなります(図2)。
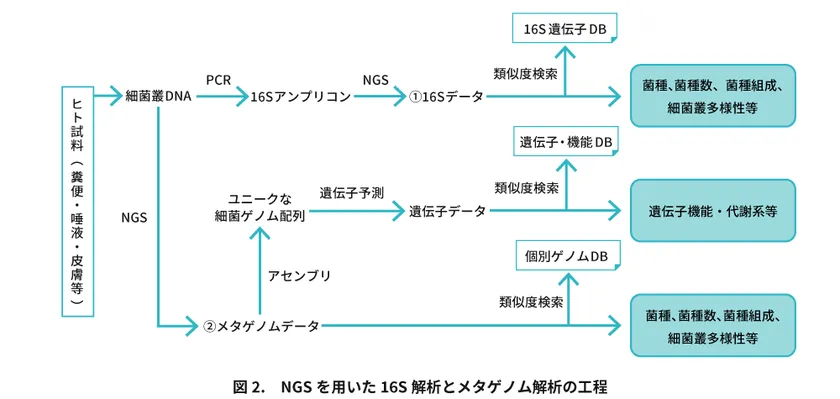
このように、長年の試行錯誤で確立された解析工程ですが、今なお技術的な改良点がいくつも残されています。
そのひとつは、第1ステップの細菌叢DNA抽出法です。
これまでに様々な細菌叢DNAの抽出法が報告され、いくつものキットが販売されてきました。
しかし、それぞれのキットや抽出法で抽出された細菌叢DNAの解析から得られる細菌叢データ(菌種や菌種組成など)が、用いたDNA抽出法間で無視できないほど異なる報告がいくつもされています。
このDNA抽出法の課題の解決なくしては、解析データとデータから導かれる解釈の信頼性が担保できません。
現在も新たなDNA抽出法の論文が発表され、新製品のキットが販売されていることそのものが、細菌叢DNA抽出法の課題が解決されていない証左とも言えます。
求められているのは、腸内に存在する様々な菌種が完全に溶菌するDNA抽出法なのです。
最近、細菌叢DNA抽出法の改良に極めてヒントになる2つの論文が報告されました。
ともに日本のグループによるもので、一つはビーズを用いた機械的溶菌法を評価した論文(Microbiome, 2021)で、もう一つは酵素を用いた溶菌法を含むいくつかの既存の溶菌法・市販キットを評価した論文(DNA Res, 2023)です。
ともに、モック・コミュニティー(20種類ほどの既知菌種からなる人工細菌叢)を用いて、モック・コミュニティーの菌種組成(正解)にもっとも類似した菌種組成となる抽出法について調べています。
結果として、前者からは機械的溶菌法のN法、P法、Q法の3つが、後者では酵素法のLA法が優位であるという結論が導かれています。
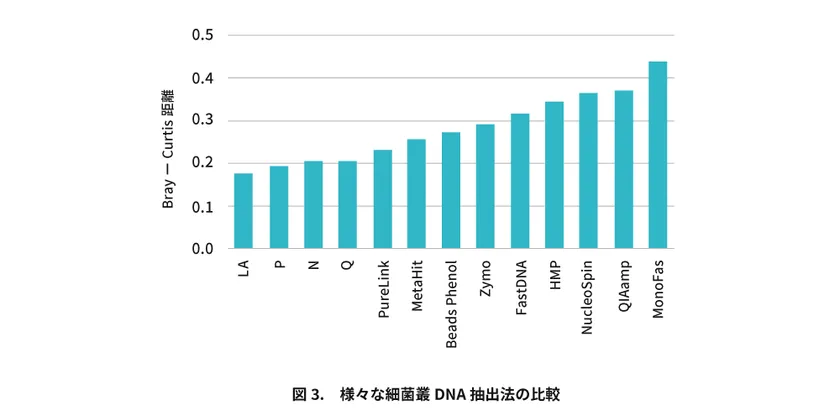
以下の図3では、2つの論文のデータを合わせて、4つの方法を含めた様々な既知法からの菌種組成とモック・コミュニティーの菌種組成間の類似性及び違いを比較しました。
LA、N、P、Q法は、他のいずれの方法よりもモック・コミュニティーの菌種組成(正解)と有意に高い類似性をもつことが分かります。
さらに、同社取締役CTO 服部 正平らは、実際の人糞便を用いて上記N、P、Q、LA法の4つを比較しました。
その結果、LA、N、P法は互換性のある高精度なヒト腸内細菌叢のDNA抽出法であることが確認されました(図4A)。
一方、Q法(Nature Biotechnology, 2017)は、今回の解析では、同じ機械的溶菌法のN法とP法よりも低い類似度を示しました(図4A)。
また、LA法がもっとも高いDNA収量をもつことも判明しました(図4B)。
これらの人糞便を用いた結果から、LA法(酵素的溶菌法)とN及びP法(いずれも機械的溶菌法)は真実の菌種組成に極めて近い菌種組成を与えることがわかりました。
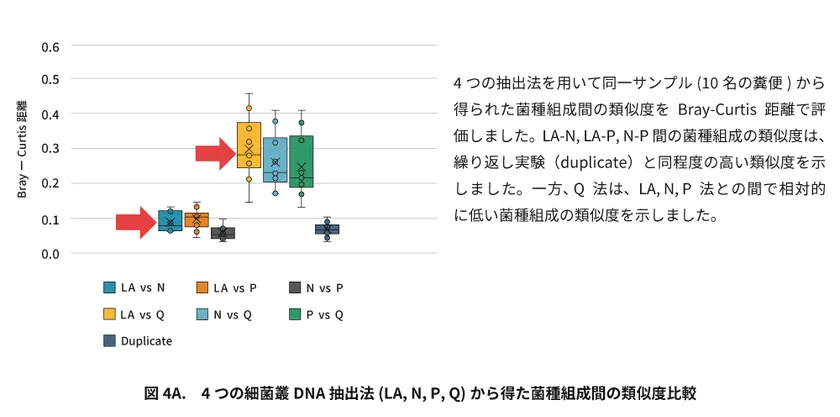
4つの抽出法を用いて同一サンプル(10名の糞便)から得られた菌種組成間の類似度をBray-Curtis距離で評価しました。
LA-N, LA-P, N-P間の菌種組成の類似度は、繰り返し実験(duplicate)と同程度の高い類似度を示しました。
一方、 Q法は、LA, N, P法との間で相対的に低い菌種組成の類似度を示しました。
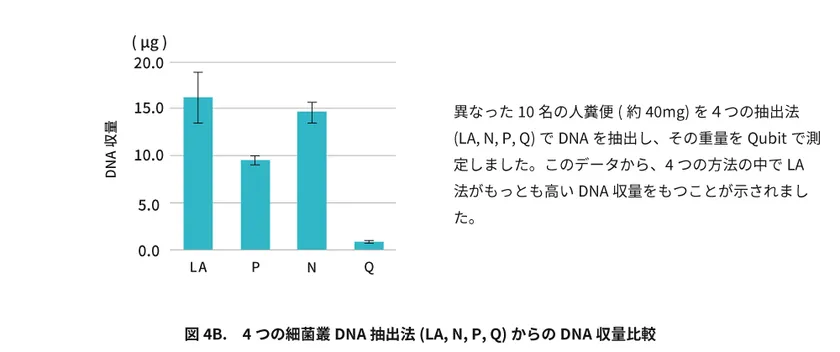
異なった10名の人糞便(約40mg)を4つの抽出法(LA, N, P, Q)でDNAを抽出し、その重量をQubitで測定しました。
このデータから、4つの方法の中でLA法がもっとも高いDNA収量をもつことが示されました。
LA法をベースにした細菌叢DNA抽出プロトコールは2013年の発表以来、すでに多くの研究プロジェクトに採用されています。
例えば、我が国初のNGSを用いた腸内細菌叢メタゲノム解析(DNA Res, 2016)や、日本最大規模の4,198名の腸内細菌叢を解析した研究(Gastroenterology. 2022)などが挙げられます。
このほか、酵素法の利点である高分子量DNAの抽出を生かしたロングリードメタゲノム解析(Microbiome, 2019)や、高いDNA収量を生かした唾液細菌叢(Nature Commun, 2021)、皮膚細菌叢(Sci Rep, 2023)などの菌数/細菌叢DNAが極めて少ないサンプルの解析にも活用されています。
■同社細菌叢DNA抽出プロトコールの特徴
・細菌種への偏りが小さい溶菌特性と高範囲な溶菌スペクトル
・高いDNA収量(数十mgの糞便、0.1mlの唾液からのNGS対応DNAの抽出)
・高分子量DNA(10kb以上の平均リード長)
・高品質DNA(NGSシークエンシングや定量PCRでの高い成功率)
・相対菌数の定量(DNA収量/糞便重量=相対菌数*)
(*同社プロトコールでは、用いた糞便量と得られるDNA収量の間にほぼ1:1の比例関係があり、個々サンプルの相対菌数の算出が可能です)
■採用事例
・日本初のNGSを用いた腸内細菌叢メタゲノム解析(DNA Res, 2016)
・日本最大規模の4,198 名の腸内細菌叢を解析(Gastroenterology. 2022)
・ロングリードメタゲノム解析(Microbiome, 2019)
・唾液細菌叢(Nature Commun, 2021)
・皮膚細菌叢 (Sci Rep, 2023)